以前、Alteryxでクラスタリング(K重心クラスター)する際に、フィールドを標準化する効果を見ましたが、今回は主成分分析ツールの「単位分散を調整する」オプションの効果を見てみたいと思います。
前提
前回と同じくscikit-learn付属のワインの区別のデータセットを使いたいと思います。こちら(uci大学)からダウンロード可能です。データの中身としては、以下のようになっています。
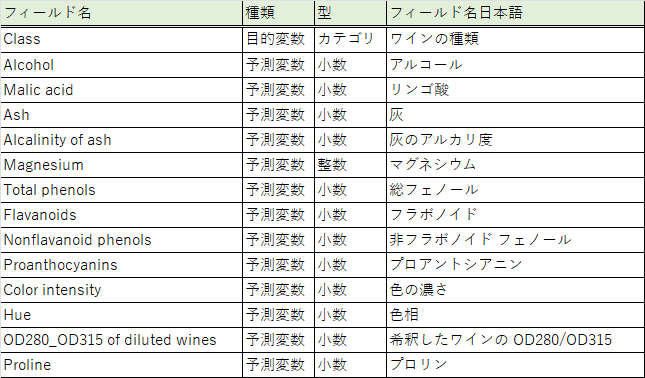
Classはクラスタリングする際の答えですが、今回は使いません。
ここで、セレクトツールで数値型にした後に主成分分析ツールに突っ込んでみましょう!

ワークフローとしては以下の通りになります。
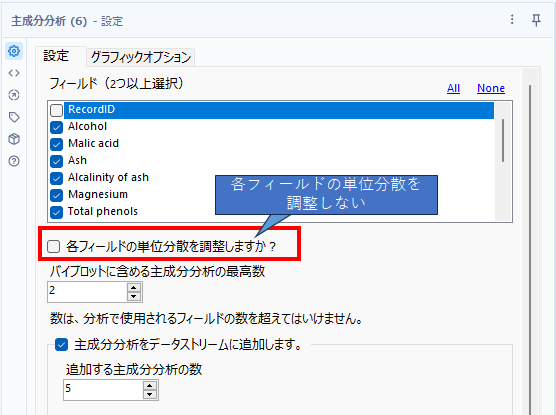
単位分散を調整しない
まず、「各フィールドの単位分散を調整しますか?」のチェックをオフにしてみたいと思います。
これで実行すると、以下のような結果が出ます。
まず、コンポーネントの要約ですが、PC1の時点で寄与率が99%にも及んでいます。
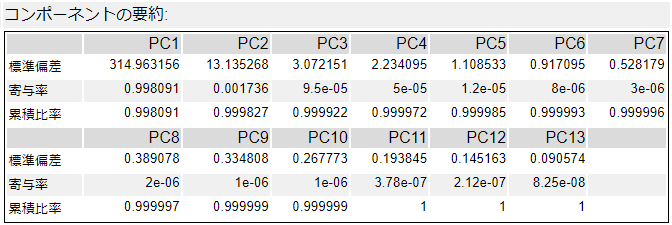
つまり、PC1でほぼすべて表現できている、ということになってしまいます。これは、Prolineという項目のスケールが278~1680となっており、他の項目であれば分散が大きくても二桁となっており、主成分分析を行う際に悪影響を与えているからです。
このようなケースでは、スクリープロットも最初の1本のみで他は不要となっていますし、バイプロットでも潰れてしまっています。
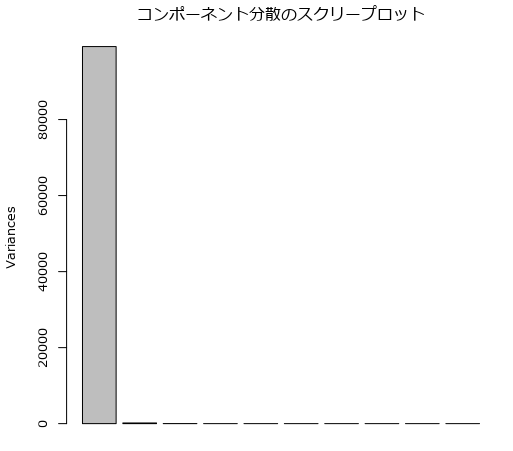
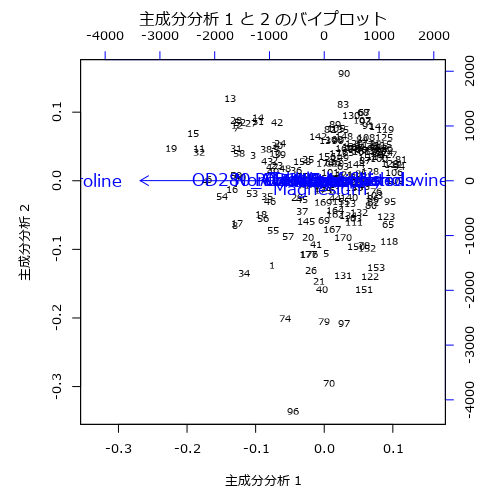
いわゆるこれは失敗パターンです。このようにスケールに偏りがある場合は、標準化などをする必要があります。
単位分散を調整する
本来、標準化を事前にしておくのですが、Alteryxの場合は、単位分散を調整するオプションが利用できます。以下のように「各フィールドの単位分散を調整しますか?」にチェックを入れるだけです。
これで実施した結果は以下のとおりです。
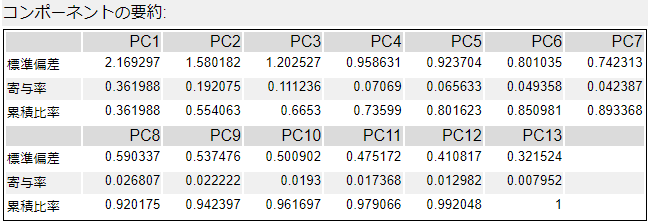
通常であれば、累積比率が60~80%を取りますが、PC5まで選択すれば80%程度になります。
スクリープロットは以下のようになっています。
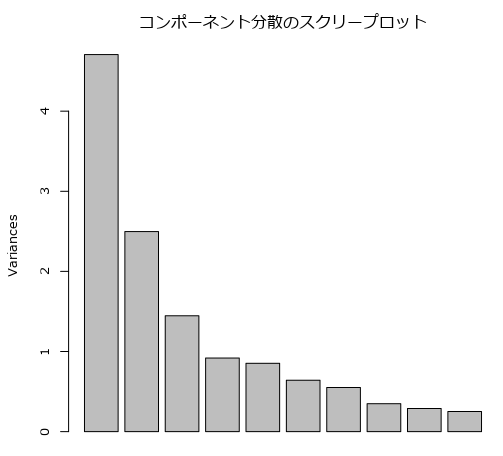
PC3くらいまででも問題なさそうにも見えます。正直、PC4、PC5の寄与率はそれほど大きくありません。
まとめ
主成分分析では、スケールがあっていないデータは事前に標準化しておく、もしくは単位分散を調整するオプションを使う必要があることがわかりました。


コメント